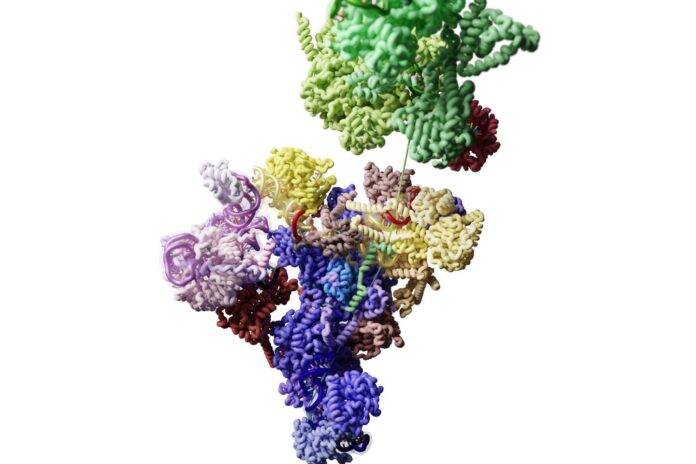
Le splicéosome. Sous ce nom abscons se cache une machinerie cruciale pour le bon fonctionnement de nos cellules. Sa mission : participer au contrôle de l’activité de nos gènes – comme de ceux de tous les organismes dits « eucaryotes », ces animaux, champignons, plantes ou levures composés de cellules dotées d’un noyau.
Dans la revue Science du 31 octobre, une équipe espagnole et américaine démonte les fins rouages de cette horlogerie biochimique. Les connaître, c’est aussi mieux comprendre un paradoxe : comment, au fil de l’évolution, les eucaryotes ont-ils pu se complexifier autant, alors même que le nombre de leurs gènes n’augmentait guère ?
« Un petit ver nématode, Caenorhabditis elegans, est pourvu de 19 000 gènes, alors que notre espèce en compte à peine plus », souligne Clément Charenton, chercheur à l’Institut de génétique et de biologie moléculaire et cellulaire (CNRS), à Strasbourg. Ainsi, nous possédons 22 000 gènes codant des protéines ; pourtant, nos cellules peuvent produire plus de 200 000 protéines différentes.
Par quel stratagème ? Ici, il nous faut revenir à une leçon de base provenant des cours de sciences de la vie et de la Terre : comment sont produites les protéines de nos cellules ? Lors d’une première étape, la séquence d’ADN d’un gène est « transcrite » en une molécule d’ARN dit « prémessager », sorte de copie de cette séquence d’ADN. Ensuite, les protéines sont formées par la traduction du message inscrit dans cet ARN, grâce à un code génétique.
Epissage alternatif
C’est entre ces deux étapes qu’opère le splicéosome. Une des clés de l’affaire se cache dans la structure des gènes : ceux-ci, le plus souvent, sont des successions de séquences d’ADN dites « codantes », les exons, et de séquences dites « non codantes », les introns. Et la molécule d’ARN prémessager est complémentaire à la fois des exons et des introns.
Mais ensuite, un étrange phénomène a lieu : les séquences d’ARN correspondant aux introns sont découpées et supprimées. Seules les séquences d’ARN correspondant aux exons, après avoir été raboutées, donneront l’ARN messager, qui sera traduit en protéines. Ce processus est nommé « épissage ». Sa découverte, en 1977, vaudra le prix Nobel de physiologie ou médecine au Britannique Richard Roberts et à l’Américain Phillip Sharp en 1993.
Au fil de l’évolution, un processus plus subtil encore, l’épissage alternatif, a été retenu par la sélection naturelle. C’est la possibilité, pour un ARN prémessager issu d’un gène donné, de subir différents épissages, conduisant à des protéines différentes.
Il vous reste 63.51% de cet article à lire. La suite est réservée aux abonnés.